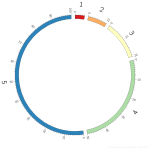
这段代码主要用于添加一个颜色图例并清除 circos 图,包含以下几个步骤: 1. `colorlegend(col, vertical = T,labels=c(1,0,-1),xlim=c(1.1,1.3),ylim=c(-0.4,0.4))` 用于添加一个颜色图例。其中 `col` 表示颜色...
”circos“ 的搜索结果
这一节的目标是画出下面的图 亮显强调 ...所谓突出标记,或者说亮显强调,多是通过大的反差明显或者符合色彩心理学的色块来将数据分组强调出来。在使用circos绘制基因组时,可以使用这一办法,将...circos流程图
考虑到circos在windows环境下安装过程比较复杂,花时间比较久,所以这里把安装需要用到的perl编译环境和circos软件,已经运行必须的模块都下载打包。但是文件过大不能上传,所以只能上传核心部分,要自己下载和运行...
label的标签有的时候没有办法完全表现出来,这个主要还是由于空间不够造成的,只看想要的基因还是很不错的,主配置文件: karyotype = data/karyotype/karyotype.human.txt chromosomes_units = 1000000 ...
写了一个小脚本用来格式化数据格式,必须有正负链的深度文件,得到gtf的基因高亮区域,并且计算gc含量和geneskew: #!/usr/bin/env perl use warnings; use strict; use Getopt::Long; ...GetOptions(\%opt, "len=s")...
axe和background属于坐标轴,前者是细线条,后者是宽线条- -!!!能这样翻译么- -!!!按照字面意思来讲,background是背景的意思,说那么多还不如画出来实在,例子添加了两个conf,但为了大家能看明白,将其写在...
用circos图展示融合基因,PPI,相关性等。
2. `circos.par(gap.degree=c(3,rep(2, nrow(cor1)-1)), start.degree = 180)` 设定 circos 图的参数,其中 `gap.degree` 定义了每个扇形之间的间隔大小,第一个元素为扇形与画布环之间的距离,后面的元素表示每个...
在绘制circos图时,文字方向与圆的切线平行,比如下图 但是,当种类过多时,可能出现文字挤在一起的情况,看不清,比如下图 有没有什么代码可以改变文字方向,使文字垂直于圆的切线 比如下图
Circos是绘制圈图的神器,在http://circos.ca/images/...Circos可以在线使用,在线使用时是把表格转为圈图,不过只允许最大75行和75列;做一些简单的示意图会比较好,最后时会介绍下在线的tableviewer的使
这段代码是用于设置 Circos 图的参数,其中 gap.after 参数是一个向量,用于指定每个区块之间的间隔大小。这个向量的长度应该是 nrow(mat) - 1 + ncol(mat) - 1 + 2,其中 nrow(mat) 和 ncol(mat) 分别是矩阵 mat 的...
#################################################################################### 一、下载安装circos及perl ##################################################################################### ...
Circos是一个JavaScript库,可以轻松地以圆形布局构建交互式图形。 它基于d3.js。 它旨在成为Circos软件的javascript版本。 您应该考虑使用Circos显示: 实体之间的关系 定期数据 从2007年(内部)到2014年(外部...
基于Perl的著名数据可视化程序Circos。 相关下载链接://download.csdn.net/download/rebelzheng/4959621?utm_source=bbsseo
这是一段 R 语言的代码,用于绘制圆形关系图,其中使用了 circos 包和 chordDiagram 函数。同时,还包括了绘制图例的代码 colorlegend。这段代码的作用是将一个相关性矩阵 cor1 转化为圆形关系图,其中每个变量对应...
According to the tutorials (http://circos.ca/documentation/tutorials/), to draw a graph using circos, at least three files needed. Download the software and install: ...
Circos.pdf 相关下载链接://download.csdn.net/download/qq_26918475/9431589?utm_source=bbsseo
interacCircos软件包设计用于生成交互式Circos图 介绍 interacCircos软件包的灵感来自 , 和 。 circosJS,BioCircos.js和NG-Circos是用JavaScript开发的,目的是提供一个框架,用于在网站上显示交互式Circos图。 ...
#../../etc/ideogram.conf the following are the default settings. Here for the convenience of comparing the default settings and the new settings with suffix * difference, some unrelated settings ar....
# /usr/bin/env python# coding=utf-8#################################### Author : yunkeli# Version : 1.0(2015/6/20)# E-mail : [email protected]###################################import osimport argp...
karyotype核型图,一般的核型图是直线模式,circos将其转换成圆形。karyotype文件格式分两部分,第一部分为染色体的总长和标识,第二部分为每条染色体的基因区域,当然第一部分是必要的。看了一下这一节的内容发现跟...
紧跟上一节的内容,主配置文件有变化,其实就是将输入文件直接格式化成颜色位置等等,没有必要一步一步的画了,文件格式如下: hs1 1298972 1300443 fill_color=blue hs1 1311738 1324571 fill_color=red,r0=0.6r,...
#/etc/circos.conf (session1.1文件注释) # karyotype路径指定:上级目录/data/karyotype.5chr.txt karyotype = ../data/karyotype.5chr.txt # chromosomes_units = 1000000 chromosomes_display_default = yes ...
Graph/Display DataCircos visualizes data in a circular layout (Galaxy Version 0.69.8+galaxy12)Circos(Krzywinski等人,2009年)是一个用于以圆形布局可视化数据的软件包。这使得Circos...
Circos提供了具体但是繁琐而且混乱的安装教程,安装、教程、答疑等label下包括了各个板块所有的内容,加之不同的操作系统的安装方式也不一样,让人看的非常疑惑。写个教程以便日后回顾。
在Notepad++下自制的circos代码编辑工具。初步试验一下效果感觉不错。这是第一个版本,如果需要改进,用户可以方便地自行完善。 相关下载链接://download.csdn.net/download/mingyuanzuo/8109749?utm_source=bbsseo
heatmap其实博主对这章有一些细节还没有完全理解,照着画倒是可以完成,但是就是心里挺不安的。。。 karyotype = data/karyotype/karyotype.human.txt chromosomes_units = 1000000 chromosomes_display_default =...
推荐文章
- 手写一个SpringMVC框架(有助于理解springMVC) 侵立删_springmvc可以用来写安卓后端吗-程序员宅基地
- 线性判别分析LDA((公式推导+举例应用))_lda推导-程序员宅基地
- C# 结构体(Struct)精讲_c# struct-程序员宅基地
- 支付宝Wap支付你了解多少?_阿里wap支付-程序员宅基地
- Java计算器编写,实现循环输入_java简易计算器可使用户多次输入-程序员宅基地
- 【多维Dij+DP】牛客小白月赛75 D-程序员宅基地
- Android之内存优化与OOM-程序员宅基地
- Azure Machine Learning - 视频AI技术_azure ai 視頻索引器-程序员宅基地
- 个人知识管理软件使用感受-程序员宅基地
- WWDC2019 ------深入理解App启动_wwdc app启动-程序员宅基地